UNIDAD 4 REPLICACIÓN DEL ADN
El proceso de replicación de ADN es el mecanismo que permite al ADN duplicarse (es decir, sintetizar una copia idéntica). De esta manera de una molécula de ADN única, se obtienen dos o más "clones" de la primera. Esta duplicación del material genético se produce de acuerdo con un mecanismo semiconservativo, lo que indica que las dos cadenas complementarias del ADN original, al separarse, sirven de molde cada una para la síntesis de una nueva cadena complementaria de la cadena molde, de forma que cada nueva doble hélice contiene una de las cadenas del ADN original. Gracias a la complementacion entre las bases que forman la secuencia de cada una de las cadenas, el ADN tiene la importante propiedad de reproducirse idénticamente, lo que permite que la información genética se transmita de una célula madre a las células hijas y es la base de la herencia del material genético.
Para que esto ocurra, la célula debe “abrir” la doble cadena de ADN en una secuencia específica denominada origen de replicación (en bacterias) o secuencia de replicación autónoma (en eucariotas) y copiar cada cadena. En la replicación participan varias enzimas. Las ADN polimerasas sintetizan una nueva cadena de ADN. Para esto utilizan como molde una de las hebras y un segmento corto de ADN, al que se le agregan los nuevos nucleótidos. Este segmento funciona como cebador (primer, en inglés). La ADN polimerasa agrega nucleótidos al extremo 3’ de la cadena en crecimiento.
4.1 Replicación del genoma procariótico
El
proceso de replicación de ADN es el mecanismo que permite al ADN
duplicarse (es decir, sintetizar una copia idéntica).
La molécula de ADN se abre como una cremallera por ruptura de
los puentes de hidrógeno entre
las bases complementarias
liberándose dos hebras y la ADN polimerasa sintetiza la mitad complementaria añadiendo
nucleótidos que se encuentran dispersos en el núcleo. De esta forma, cada nueva molécula es idéntica a la molécula de ADN inicial.
La
replicacion en los organismos procariotes es mas lento que el de los
eucariotas, a pesar de tener un solo centro de origen y tener menos problemas
con el superenrollamiento.
Tiene
la polimerasa III, 7 subunidades, la direccion de replicacion en la cadena
continua es 5 prima 3 prima, la replicacion es bidireccional, presenta un
centro de replicacion, que van a formar 2 horquillas,empieza con los
nucleotidos adenina-timina, por tener que gastar menos energia para romper los
puentes de hidrogeno de las cadenas(helicasa).
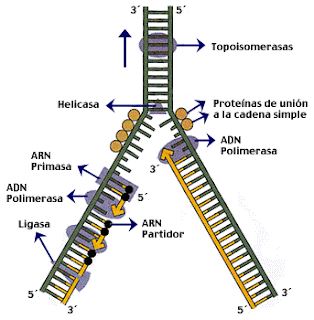
Proceso de replicación:

- La topoisomerasa impide que el ADN se enrede debido al
superenrollamiento producido por la separación de la doble hélice,es
decir, la hace menos densa y la relaja para que se puedan dividir
las cadenas.esta es la primera enzima que actúa en la replicación.
- centro de origen: al irse desenrollando
las cadenas, se van formando como un espacio como si fuera una burbuja, en
el cual se inicia la replicación, separándose las cadenas en un centro de
origen formándose dos horquillas.
- La helicasa rompe los puentes de hidrógeno de la doble
hélice permitiendo el avance de la horquilla de replicación. esta enzima
es la que va a dividir la doble hélice de la cadena.
- La helicasa rompe los puentes de hidrógeno de la doble
hélice permitiendo el avance de la horquilla de replicación. esta enzima
es la que va a dividir la doble hélice de la cadena.
- Las proteínas SSB se unen
la hebra discontínua de ADN, impidiendo que ésta se una consigo misma.
estas proteinas impiden que se vuelvan a unir las cadenas separadas.
- La ADN polimerasa sintetiza la cadena complementaria de forma
continua en la hebra adelantada y de forma discontinua en la hebra
rezagada.la DNA polimerasa III es la encargada de sintetizar la cadena
continua y la ADN polimerasa I se encarga de sintetizar la cadena
resagada. ya que en la cadena resagada la ADN polimerasa no puede
sintetizar por completo la cadena y por eso se forman unos fragmentos
llamados fragmentos de okasaki, que después van a actuar sobre estos la
ADN polimerasa I, rellenando los espacios dejados por la ADN polimerasa
III, para ir sintetizando los nuevos nucleotidos de la cadena nueva.
- La ARN primasa sintetiza el cebador de ARN necesario para la
síntesis de la cadena complementaria a la cadena rezagada. es decir, esta
estructura va a permitir que la ADN polimerasa actué para sintetizar las
nuevas cadenas, ya que en el va a empezar a sintetizar.
- La ADN ligasa une los fragmentos de Okazaki con la ADN
polimerasa III, para que la cadena este sintetizada completamente.
- El cebador: son pequeñas unidades de RNA que se unen a los
fragmentos para que la ADN polimerasa reconozca donde debe unirse.
4.2 REPLICACIÓN DEL GENOMA EUCARIÓTICO
 El mecanismo para
la síntesis del DNA en eucariontes probablemente es similar al proceso en
procariontes.
El mecanismo para
la síntesis del DNA en eucariontes probablemente es similar al proceso en
procariontes.
4.3. CONTROL DE LA REPLICACIÓN
El proceso
resultante de la duplicación de ADN se conoce como división celular, la cual se
ha estudiado a nivel citológico estableciéndose ciertas pautas cubiertas por lo
que se conoce como ciclo celular.
Las células de los
distintos organismos pasan durante su vida por distintos períodos, cada uno de
ellos característico y claramente diferenciado.
Cada tipo celular cumple
con sus funciones específicas durante la mayor parte de su vida, creciendo
gracias a la asimilación de materiales provenientes de su ambiente y con ellos
sintetiza nuevas moléculas por medio de complejos procesos regulados por su
material genético.
 Cuando una célula aumenta
hasta llegar a un determinado tamaño, su eficiencia metabólica se torna
crítica, entonces se divide. En los organismos pluricelulares, se produce un
crecimiento a partir de una célula (huevo o cigoto) como así también se aumenta
la masa tisular y se reparan los tejidos lesionados o desgastados, por aumento
del número de células.
Cuando una célula aumenta
hasta llegar a un determinado tamaño, su eficiencia metabólica se torna
crítica, entonces se divide. En los organismos pluricelulares, se produce un
crecimiento a partir de una célula (huevo o cigoto) como así también se aumenta
la masa tisular y se reparan los tejidos lesionados o desgastados, por aumento
del número de células.
Las nuevas células
originadas en esta división poseen una estructura y función similares a las
células progenitoras, o bien derivadas de ellas.
El cromosoma de la
bacteria intestinal Escherichia coli es único, circular y contiene cerca de 4.7
millones de pares de bases. Tiene cerca de 1 mm de longitud pero solo 2 nm de
ancho. El cromosoma se replica produciendo una figura que asemeja a la letra
griega theta.
El promotor es la
parte del ADN en donde se pega la ARN polimerasa antes de abrir el segmento de
ADN a ser transcripto.
Un segmento del
ADN que codifica para un polipéptido específico se conoce como un gen
estructural . Escherichia coli puede sintetizar 1700 enzimas, por lo tanto esta
pequeña bacteria tiene genes para 1700 mARN.
 La Reacción en
Cadena de la Polimerasa (PCR) es una técnica de laboratorio que permite la
copiain vitro de
secuencias específicas de DNA y que ha revolucionado el campo de la Biología
Molecular. Conceptualmente, la PCR es una técnica sencilla, consiste en la
separación por calor de las dos cadenas del DNA que se quiere amplificar, y su
copia simultánea a partir de un punto, determinado por un fragmento de DNA
artificial llamado cebador, mediante la acción de una enzima denominada DNA
polimerasa. El resultado es la duplicación del número de moléculas de una
secuencia concreta de DNA. Este proceso se repite un número determinado de
veces o ciclos, generalmente 30, y se consigue un aumento o amplificación
exponencial del número de copias del fragmento de DNA molde. Por tanto, si
partimos de una molécula única de DNA en la muestra inicial, y en cada ciclo se
duplica el número de moléculas, teóricamente, al final de los 30 ciclos, se
obtendrán 230moléculas idénticas, es decir 1073741824 moléculas.
La Reacción en
Cadena de la Polimerasa (PCR) es una técnica de laboratorio que permite la
copiain vitro de
secuencias específicas de DNA y que ha revolucionado el campo de la Biología
Molecular. Conceptualmente, la PCR es una técnica sencilla, consiste en la
separación por calor de las dos cadenas del DNA que se quiere amplificar, y su
copia simultánea a partir de un punto, determinado por un fragmento de DNA
artificial llamado cebador, mediante la acción de una enzima denominada DNA
polimerasa. El resultado es la duplicación del número de moléculas de una
secuencia concreta de DNA. Este proceso se repite un número determinado de
veces o ciclos, generalmente 30, y se consigue un aumento o amplificación
exponencial del número de copias del fragmento de DNA molde. Por tanto, si
partimos de una molécula única de DNA en la muestra inicial, y en cada ciclo se
duplica el número de moléculas, teóricamente, al final de los 30 ciclos, se
obtendrán 230moléculas idénticas, es decir 1073741824 moléculas.
La gran ventaja de la PCR es que amplifica únicamente el fragmento de
DNA que queremos aunque esté en cantidades mínimas (alta sensibilidad) o en
presencia de grandes cantidades de otros DNA semejantes (alta especificidad).
La reacción es eficaz incluso si se parte de muestras de DNA muy poco
purificadas, en presencia de otros componentes.
La PCR se ha convertido en una
herramienta fundamental en campos tan diversos como la investigación (clonado
de genes, detección de clones recombinantes, estudio de DNA de fósiles),
medicina forense y criminalística (determinación de la paternidad, identificación
de individuos, identificación sospechoso/evidencia en casos de asesinato,
violación, etc.), sanidad animal y mejora genética (detección de enfermedades
genéticas e infecciosas, pureza de razas, etc.) y medicina (diagnóstico de
enfermedades). La PCR fue desarrollada por Kary Mullis (Saiki et al, 1985;
Mullis, 1990) en 1985, utilizando como DNA polimerasa el fragmento Klenow de la
DNA polimerasa I de E. coli y lo llevó a cabo cambiando los tubos de reacción
en cada ciclo, entre dos baños que estaban a 94 ºC (desnaturalización) y 37 ºC
(hibridación y extensión del cebador). Debido a que esta enzima se
desnaturaliza a temperaturas superiores a 37 ºC, era necesario añadirla en cada
uno de los ciclos tras el paso de desnaturalización, lo que convertía a la PCR
en una técnica tediosa y costosa
4.4.1
SECUENCIACIÓN DEL ADN
La secuenciación de
ADN es un conjunto
de métodos y técnicas bioquímicas cuya finalidad es la
determinación del orden de
los nucleótidos (A, C, G y T) en
un oligonucleótido de ADN. La secuencia de ADN constituye la
información genética heredable del núcleo celular, los plásmidos,
la mitocondria y cloroplastos (En plantas) que forman la
base de los programas de desarrollo de los seres vivos. Así pues, determinar la
secuencia de ADN es útil en el estudio de la investigación básica de los
procesos biológicos fundamentales, así como en campos aplicados, como la
investigación forense. El desarrollo de la secuenciación del ADN ha
acelerado significativamente la investigación y los descubrimientos
en biología. Las técnicas actuales permiten realizar esta secuenciación a
gran velocidad, lo cual ha sido de gran importancia para proyectos de
secuenciación a gran escala como el Proyecto Genoma Humano. Otros
proyectos relacionados, en ocasiones fruto de la colaboración de científicos a
escala mundial, han establecido la secuencia completa de ADN de
muchos genomas de animales, plantas y microorganismos.
Una secuencia
de ADN o secuencia genética es una sucesión de letras
representando la estructura primaria de una molécula real o
hipotética de ADN o banda, con la capacidad de
transportar información.
Las posibles
letras son A, C, G, y T, que
simbolizan las cuatro subunidades de nucleótidos de una banda ADN
- adenina, citosina,guanina, timina, que son bases
covalentemente ligadas a cadenas fosfóricas. En el típico caso, las secuencias
se presentan pegadas unas a las otras, sin espacios, como en la secuencia
AAAGTCTGAC, yendo de 5' a 3' de izq. a derecha.
Una sucesión de
cualquier número de nucleótidos mayor a cuatro es pasible de llamarse una
secuencia. En relación a su función biológica, que puede depender del contexto,
una secuencia puede tener sentido o antisentido, y ser
tanto codificante o no codificante. Las secuencias de ADN pueden
contener "ADN no codificante."
Las secuencias
pueden derivarse de material biológico de descarte a través del proceso
de secuenciación de ADN.
En algunos casos
especiales, las letras seguidas de A, T, C, y G se presentan en una secuencia.
Esas letras representan ambiguedad. De todas las moléculas muestreadas, hay más
de una clase de nucleótidos en esa posición. Las reglas de la Unión
Internacional de Química Pura y Aplicada (IUPAC) son las que siguen:
A = adenina
C = citosina
G = guanina
T = timina
R = G A (purina)
Y = T C (pirimidina)
K = G T (keto)
M = A C (amino)
S = G C (enlaces fuertes)
W = A T (enlaces débiles)
B = G T C (todos y A)
D = G A T (todos y C)
H = A C T (todos y G)
V = G C A (todos y T)
N = A G C T (cualquiera)
BIBLIOGRAFÍA:
http://quimicoclinico.wordpress.com/2008/03/27/anticuerpos-contra-vih/
Sanger F, Coulson AR. A rapid method for determining
sequences in DNA by primed synthesis with DNA polymerase. J Mol Biol. 1975
Mayo 25;94(3):441–448
http://www.efn.uncor.edu/dep/biologia/intrbiol/adntema3.htm
http://es.wikipedia.org/ imagenes


No hay comentarios:
Publicar un comentario